— A Course for Graduates and Junior/Senior Undergraduates (本硕贯通课程-叁年。)
《生物文本挖掘和知识发现概论》(课程代码:317210004009) 自2016年起向研究生开设。2020年尝试性面向生物信息专业本科生讲授,课程名为《自然语言处理与知识发现》(课程代码3173009011)。2021年,本课程被设置为本硕贯通课程。
因跟随生物信息专业2023年度的本科培养方案调整,自今年(2023年)起,授课学期更换为秋季。
今年选课的同学为生物信息2021级本科生10名,研究生12名(生物信息学和生物/医药方向)。
Apply the NLP ideas in Biomedicine Texts
欢迎选课
Welcome to the course: “BioNLP and Knowledge Discovery”
这门课程源于2016年起开设的研究生《生物文本挖掘与知识发现概论》,每年春季授课。2020年对生物信息专业本科生增开。2021年起,作为本硕贯通课程开设。课程大纲和课程相关资源请在本页获取。
我为什么要选(不要选)这门课程?
学生评语(2023年春):“老师的课程内容非常硬核,需要付出大量的精力才能完成,但是从中我获得了很多思维的启发,让我获得了初步的科研思维。提个小建议,在课程初期就要求完成短文,但在课程极后期才在课上讲解使用pubtator下载数据的方式,导致初期短文进度艰难,建议斟酌过程考核和课程安排,祝课程未来更好!”(注:根据此条建议,将《依存关系和深层语义》和《PubMed实体识别和Shell编程》进行了前后调换。)
学生评语(2023年春):“非常Push但是能够学到很多东西,收获远大于学习过程的艰难。”
学生评语(2023年春):“课上讲的部分东西对要写的大论文联系不够多,对代码的讲解较少,课程很难!”
学生评语(2022年):“课程节奏紧张,任务较难,但老师讲解十分耐心,积极回应积极帮助同学们的各种疑问。”
学生评语(2021年):“老师讲的很好,让我了解到一些以前接触较少得东西,十分有用,但是对于我这样代码基础较弱的来说还是有一定的挑战性。”
学生评语(2021年): “老师注重启发学生思考,从不同的角度引导学生,培养学生的科研思维。由于老师非常负责对学生要求很高,期望也很高,对学生较为严厉,缺少对学生的鼓励,学生课业压力较大。” (注:根据此条建议,主动调高了亲和力属性值。)
Discover The Syllabuse
发现课程
一、日程安排
| 日期 | 内容 |
| 11, Sep | ▩ Ch0.《课程概论》 ▩ Ch1.《生物医药自然语言处理BioNLP的基本问题》(Slides) ▩ 课程大论文数据发布:作物文献(~40k PubMed abs) / 疾病文献(Long Covid) |
| 18, Sep | ▩ 论文阅读. Chen, Qingyu, et al. “Multi-label classification for biomedical literature: an overview of the BioCreative VII LitCovid Track for COVID-19 literature topic annotations.” Database 2022 (2022): baac069. (论文Link) ▩ Ch2.《词汇计算基础》(Slides) |
| 25, Sep | ▩ Ch3. 《NLTK文本预处理:分词、分句、词频、POS、近似词汇》(Slides) ▩ 专题1:《作物/疾病文献的知识挖掘目标》 |
| 27, Sep 实验课 | ◎ 实验任务一、《基本词汇计算》 |
| 9, Oct | ▩ Ch4. 《PubMed实体识别和Shell编程》(Slides) |
| 11, Oct 实验课 | ◎ 实验任务二、《词云计算》 附加资料:《使用E-utilities从PubMed批量获取文献摘要》by 刘雅文 |
| 16, Oct | ▩ Ch5. 《本体和本体富集 (GO, HPO 和PTO)》(Slides) |
| 18, Oct 实验课 | (课程大论文短文提交日) ◎ Quiz A:算法理论考题 ◎ 实验任务三、《PubMed文献实体识别》 ◎ 专题2:《作物/疾病文献的词汇分析》 |
| 23, Oct | ▩ Ch6. 《依存关系和深层语义》(Slides) |
| 25, Oct 实验课 | (课程大论文长文初稿提交日) ◎ Quiz B:代码考核 ◎ 实验任务四、《本体富集分析》 |
| 30, Oct | ▩ Ch7. 《语义和嵌入计算》(Slides) 延伸了解:《讨论下GPT Family》Mar 2023 |
| 6, Nov | (课程大论文长文修订稿提交日) ▩ Quiz C:《作物、疾病文献挖掘》小组汇报 专题3:《生物文本挖掘和知识图谱》 |
| 15, Nov 实验课 | ◎ 随堂考查 ◎ 实验任务五、《Word2Vec语义嵌入》 |
| 22, Nov | (课程大论文最终稿提交日) 请将pdf文件提交到课程微信群 截止时间:11:59 pm, 22, Nov. |
days
hours minutes seconds
until
课程大论文最终稿(电子版)
二、ITC实验安排
◎ 实验二 10/11/2023(第五周)
| 名称 | 《词云计算》 |
| 时长 | 2节课 |
| 学习目的 | R调包 |
| 数据 | PubMed文献数据 |
| 实现方式 | Python |
| 作业 | 实验材料:作物文献(~40k PubMed abs) / 疾病文献(Long Covid) • 基本要求: —复现Python命令,绘制词云图,截图上交 • 进阶要求(Optional): —词云图能体现热点词汇/研究趋势。 |
◎ 实验三 10/18/2023(第六周)
| 名称 | 《PubMed文献实体识别》 |
| 时长 | 4节课 |
| 学习目的 | Shell脚本抓取PubTator网页,对所抓取的海量文本的分析 |
| 数据 | Shell编程,API抓取 |
| 实现方式 | Terminal终端 + Jupyter |
| 作业 | • 基本要求: —复现Shell代码,获得目标文献pmid,获取部分PubTator标注文本,截图上交。 • 进阶要求(Optional): —依据Jupyter代码部分的关键词分析代码,对PubTator文本进行初步筛选,对结果进行分析和解读 |
◎ 实验四 10/25/2023(第七周)
| 名称 | 《本体富集分析》 |
| 时长 | 4节课 |
| 学习目的 | 一个具体的算法的调包实现 |
| 数据 | 自动下载的数据 |
| 实现方式 | R Studio 或 Python |
| 作业 | • 基本要求: —复现R代码,获得富集分析结果,截图上交。 • 进阶要求(Optional): —从前期文本挖掘实验中挑选若干基因,获得富集分析结果,对结果进行分析和解读。 —实现HPO富集的Python实现,参考GOATOOLS GitHub项目。 |
三、课程大论文安排
| 名称 | 《作物/疾病文献挖掘和知识发现》 |
| 时长 | 整个学期 |
| 要求 | • 课程大论文为必选项; • 自主挑选队员组队,每组3人; • 一位同学可以参加多组; • 组员名单和题目在每次论文提交点均可进行调整; • 组员排名不分先后,请在论文中清晰介绍各人分工和贡献。 |
| 数据 | 使用指定数据: |
| 论文格式 | 论文无特别格式要求。 |
| 提交时间 | 依时间点,逐次提交短文,长文初稿,修改稿,终稿。 |
四、课程考核
- 平时 60%
- 实验报告 20%
- 三次课堂测验 20% (与课程大论文进度考察有关)
- 课堂表现 20% (与课程大论文进度考察有关)
- 期末考试 40%
五、课程讨论进程
♥ 词汇计算部分-课中和课后讨论 (2023-09-18)
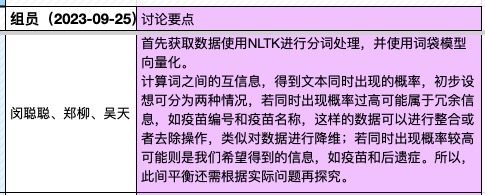
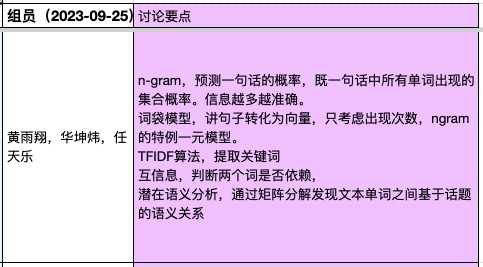
♥ 知识发现的常见研究范式-课中讨论,课程论文Idea (2023-09-25)
♥ 课程论文-短文思路 (2023-10-09)
♥ 课程论文-短文评阅 (2023-10-18)
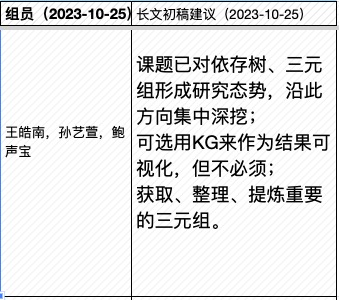
♥ 课程论文-长文初稿评阅 (2023-10-25)





♥ 课程论文-长文修订稿 (2023-11-06)
Course Resources and External Links
课程资源和外部链接
六、推荐文献-课前阅读
☁ 1. 【了解近十年来BioNLP领域的前沿问题】Zhao, S., Su, C., Lu, Z., & Wang, F. (2021). Recent advances in biomedical literature mining. Briefings in Bioinformatics, 22(3), bbaa057. (论文链接)
☁ 2. 【了解一个文本/主题分类的场景】 Chen, Qingyu, et al. “Multi-label classification for biomedical literature: an overview of the BioCreative VII LitCovid Track for COVID-19 literature topic annotations.” Database 2022 (2022): baac069. (论文链接)
☁ 3. 【了解一个知名的生物医药专属的语料库资源】Kim, J. D., Ohta, T., & Tsujii, J. I. (2008). Corpus annotation for mining biomedical events from literature. BMC bioinformatics, 9(1), 1-25. (论文链接)
☁ 4.【了解一个本体资源与生物信息学知识发现的结合案例】Wang, S., Meng, X., Wang, Y., Liu, Y., & Xia, J. (2019). HPO-Shuffle: an associated gene prioritization strategy and its application in drug repurposing for the treatment of canine epilepsy. Bioscience reports, 39(9). (论文链接)
☁ 5. 【了解一个流行的全PubMed范围的生物医学知识标注资源】Wei, C. H., Allot, A., Leaman, R., & Lu, Z. (2019). PubTator central: automated concept annotation for biomedical full text articles. Nucleic acids research, 47(W1), W587-W593.(论文链接)
☁ 6.【了解斯坦福语法依存树结构】De Marneffe, M. C., & Manning, C. D. (2008). Stanford typed dependencies manual (pp. 338-345). Technical report, Stanford University.(文献链接)
所有课程
♥ 回到 我的课程列表和逻辑关系图

Find us!
找到我们!
课后答疑
办公室: 逸夫楼C610

助教:贺芷涵
Course for BioNLP
学科交叉,融会贯通,学好BioNLP.
Course Hours
See jw.hzau.edu.cn
Office
C610, Yifu bldg
Contact me
xiajingbo.math@gmail.com